来源:
在活细胞内,蛋白质和其他分子通常非常紧凑地堆积在一起。因为荧光标记无法嵌入分子之间而使它们可见,所以这些密集的簇很难成像。据《自然·生物医学工程》杂志报道,美国麻省理工学院的研究人员开发出一种新的方法来克服这一限制,使这些“看不见”的分子变得可见。该技术通过在标记之前扩大细胞或组织样本,降低分子的拥挤度,从而使它们更容易被荧光标记。这种方法以扩张显微镜技术为基础,可以使科研人员观察到以前从未见过的分子和细胞结构。
麻省理工学院的生物工程、大脑和认知科学教授Edward Boyden表示:“扩张过程将揭示许多新的生物学发现。因为如果生物学家和临床医生在研究大脑或其他生物样本中的蛋白质时,一直以常规方式对其进行标记的话,他们可能会遗漏某些现象。”
Boyden 及其同事通过这项技术可以对神经元突触中发现的纳米结构进行成像。而且他们还对与阿尔茨海默病相关的β淀粉样蛋白斑块的结构进行了更详细的成像。
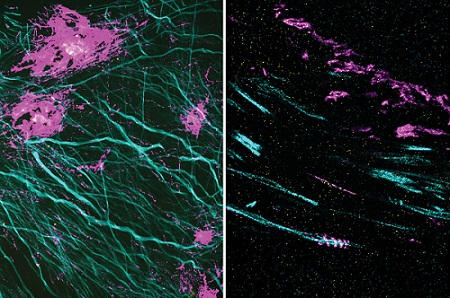
图 洋红色标记是β淀粉样蛋白纳米结构,通过扩张后染色显示。左图显示了β淀粉样蛋白纳米团簇的线状图案,右图显示了β淀粉样蛋白的螺旋结构。
通常来讲,对细胞内的特定蛋白质或其他分子进行成像需要用与靶标结合的抗体携带的荧光标签进行标记。由于抗体长约 10 nm,而典型的细胞蛋白直径通常约为 2 至 5 nm,因此如果靶蛋白堆积得太密,抗体就无法与之结合。这一直是传统成像以及 Boyden 于 2015 年首次开发的扩张显微镜的障碍。在那个时候,研究人员在扩张组织之前就要将荧光标记附加到目标分子上。提前进行标记的部分原因是研究人员必须使用一种酶来切割样品中的蛋白质,以便扩大组织。
为了克服这个障碍,研究人员必须找到一种方法,可在扩大组织的同时保持蛋白质的完整。他们决定使用热量而不是酶来软化组织,使其膨胀二十倍而不被破坏。之后,分离出的蛋白质可以用荧光标签进行标记。
有了许多可用于标记的蛋白质,研究人员就能够识别突触内的微小细胞结构,即紧密堆积着蛋白质的神经元之间的连接。他们对七种不同的突触蛋白进行了标记和成像,这使得他们能够详细地观察到由钙通道与其他突触蛋白排列而成的“纳米柱”。这些“纳米柱”被认为有助于提高突触交流效率,于 2016 年在马里兰大学教授、该研究的主要成员之一 Thomas Blanpied 的实验室中首次发现。
麻省理工学院博士后研究员 Jinyoung Kang 认为,该项技术可用于解决许多与神经退行性疾病有关的突触蛋白功能障碍的生物学问题。毕竟到目前为止,还没有能够有效地使突触可视化的工具。
研究人员还使用这项新技术对阿尔茨海默病患者大脑中形成斑块的 β-淀粉样肽进行成像。 利用小鼠的脑组织,研究人员发现,β淀粉样蛋白形成了以前从未见过的周期性纳米簇,这些β淀粉样蛋白簇还包括钾通道。此外,他们还发现了沿着轴突形成螺旋结构的β淀粉样蛋白分子。
Boyden 和他的团队现在正在与其他实验室合作研究细胞结构,比如与帕金森氏症和其他疾病相关的蛋白质聚集体。在其他项目中,他们正在研究感染与大脑衰老有关的细胞和分子的病原体。Boyden说,这些研究的初步结果也揭示了新的细胞结构。“你会看到一次又一次令人震惊的新突破,通过这项技术,我们得以看到许多在经典未扩展染色方面未曾发现的现象。”
他们计划改进这项技术,以便一次可以成像多达 20 种蛋白质,或者有希望可以用于人体组织样本。另一方面,Sarkar 和他的团队正在开发可以分布在大脑中的微型无线供电纳米电子设备。他们计划使用扩张显示来集成这些设备。 “这可以将纳米电子学的智能与扩张技术的纳米技术能力相结合,从而对大脑的功能和结构进行综合研究,”Sarkar 说道。
该研究发表在 Nature Biomedical Engineering (www.doi.org/10.1038/s41551-022-00912-3) 。
